Arterioveneuze malformaties (AVM’s) zijn abnormale verbindingen tussen arteriën en venen, die niet onderling verbonden zijn met een normaal capillair bed. Door de hoge flow van de arterie in de vene ontstaat er een hoge veneuze druk, die kan leiden tot een ruptuur met mogelijk dodelijke bloedingen tot gevolg. Cerebrale AVM’s komen voor in 1.12 tot 1.42 gevallen per 100.000 persoonsjaren en hebben een bloedingsrisico van ongeveer 3.0% per jaar [1]. Micro-AVM’s zijn AVM’s met een diameter van minder dan 1,0 cm en vertegenwoordigen hiermee 8% van alle cerebrale AVM’s [2]. Ondanks de geringe grootte zijn mAVM’s verantwoordelijk voor een groot aantal klinisch significante bloedingen in jongvolwassenen [3].
In patiënten met een mogelijke AVM wordt als beeldvorming Magnetic Resonance Imaging (MRI) en digitale substractie angiografie (DSA) ingezet. mAVM’s zijn vaak niet zichtbaar op 3D MRI-beelden, maar wel op 2D DSA [4]. Deze DSA bestaat uit een coronaal en sagittaal beeld, waardoor de AVM in twee richtingen gelokaliseerd kan worden. Voor preoperatieve planning is het belangrijk de exacte 3D locatie van het AVM te weten [5,6]. Om deze locatie te visualiseren is fusie van de DSA met de MRI noodzakelijk [7].
In de literatuur zijn verschillende methoden beschreven voor de fusie van 2D beelden met MRI beelden, welke te zijn onderverdelen in de ‘intensity-based’ en de ‘feature-based’ methoden. Intensity-based methoden maken gebruik van de overeenkomst tussen de voxel-intensiteiten van beelden van verschillende modaliteiten [8]. In de feature-based methode worden primitieve eigenschappen van de beelden, zoals scherpe randen of grote oppervlakten met dezelfde eigenschappen met elkaar gecombineerd [9]. In de huidige studies waarbij de fusie van de cerebrale vaten is beschreven, werd als 3D beeld gebruikt gemaakt van Magnetic Resonance Angiografie (MRA), waarbij de vaten makkelijk te herkennen zijn. Omdat in deze studie gebruik wordt gemaakt van reguliere MRI zijn eerder beschreven methodes niet toereikend.
Het doel van deze studie is het creëren van een software tool die automatisch een AVM lokaliseert en visualiseert op MRI. Hiervoor zijn de coördinaten van de AVM op een sagittale en coronale DSA nodig als input.
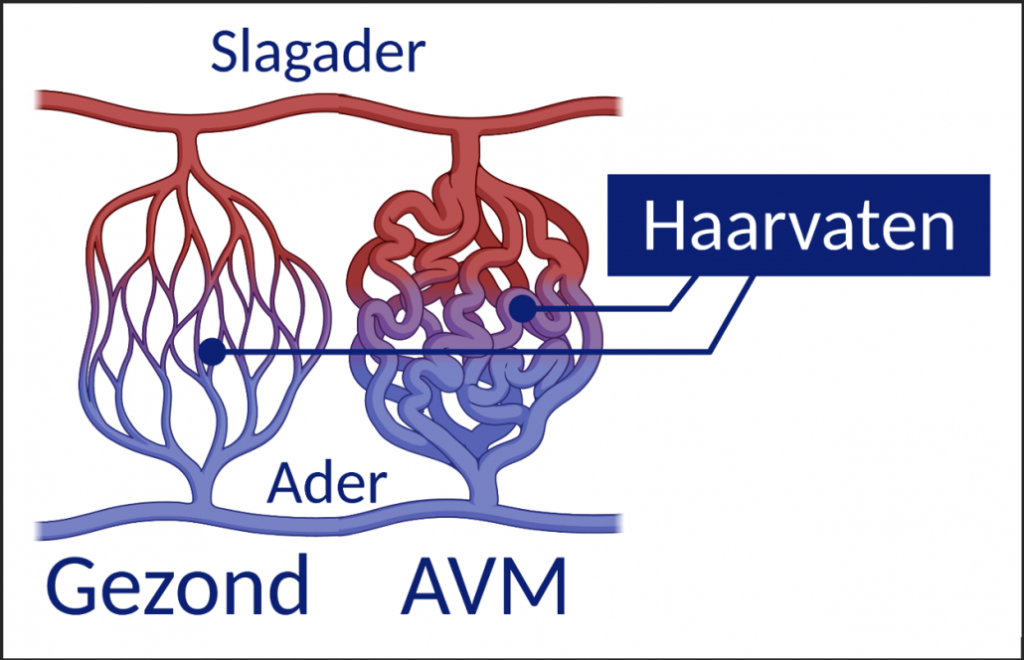
Links: normale verbinding tussen arterie en vene. Rechts: AVM [11]
Methodologie
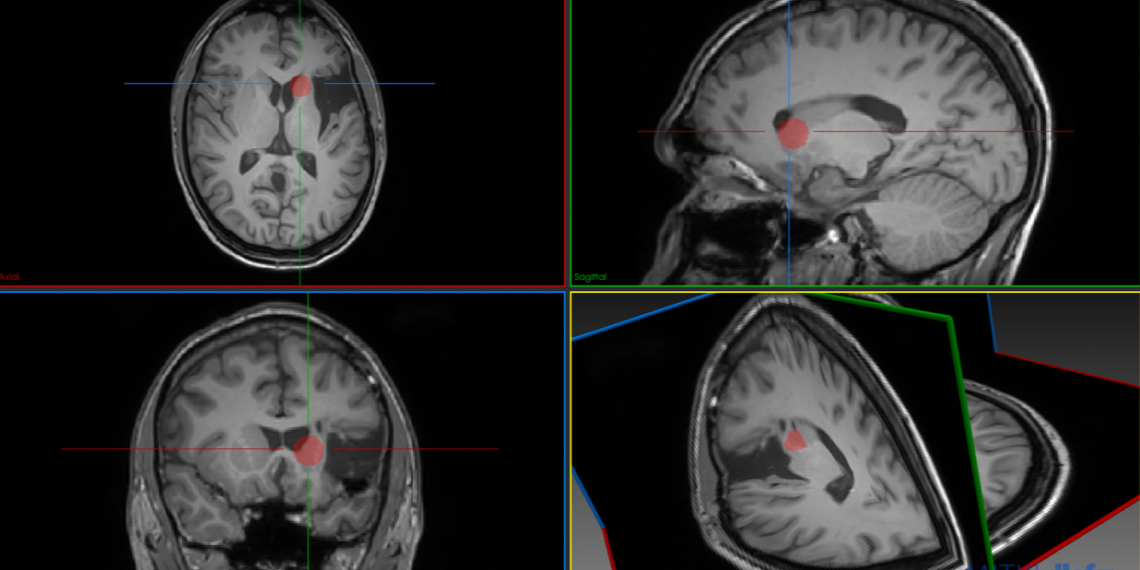
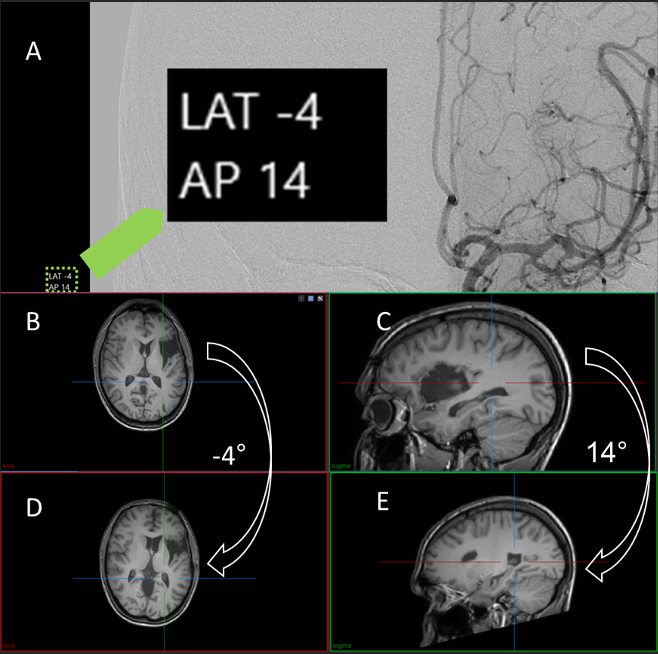
MRI en DSA van patiënten met mAVM’s werden geanalyseerd in Matlab R2020b, waarbij gebruik werd gemaakt van de beeldregistratie-software Elastix [10]. Als eerste stap werden alle DICOM-slices van de MRI samengevoegd tot één 3D-volume. Vervolgens werden de hoeken waaronder de angiografie gemaakt was afgelezen uit de DICOM-informatie van de DSA. Om ervoor te zorgen dat de MRI in hetzelfde coördinatensysteem stond als de DSA werd op de originele MRI een rotatie toegepast, waardoor er één MRI ontstond in hetzelfde assenstelsel als de coronale DSA, en één MRI in hetzelfde assenstelsel als de sagittale DSA (Figuur 1). Vervolgens werd uit deze MRI’s respectievelijk de coronale en sagittale slice met de grootste oppervlak geselecteerd en verder geanalyseerd.
Segmentatie
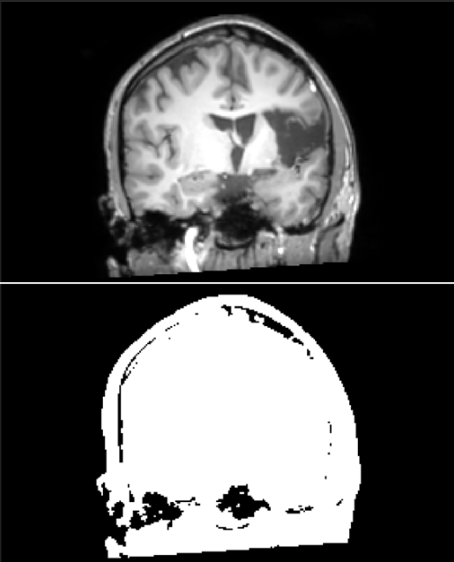
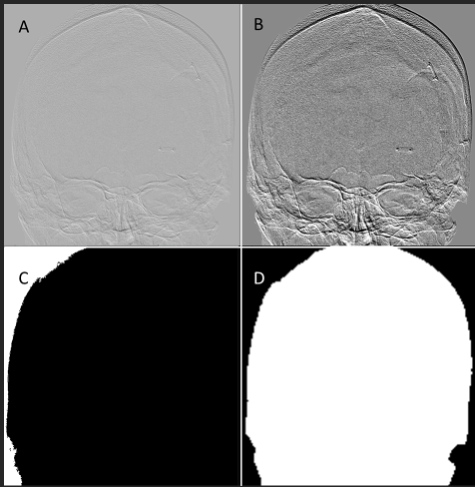
Op deze geselecteerde MRI-slices werd met een eenvoudige threshold-based segmentatie de contour van het hoofd gesegmenteerd (Figuur 2). Ook op de DSA werd het hoofd gesegmenteerd. Hiervoor werd de tweede slice van de angiografie sequentie gebruikt. Op deze slice was nog geen contrast zichtbaar, maar zijn de contouren van het hoofd wel duidelijk te onderscheiden. Met een region-growing algoritme in de vier uiterste hoeken van dit DSA beeld werd de achtergrond gesegmenteerd. Door al deze hoeken te classificeren als achtergrond bleef de contour van de schedel over als segmentatie (Figuur 3).
Lokalisatie
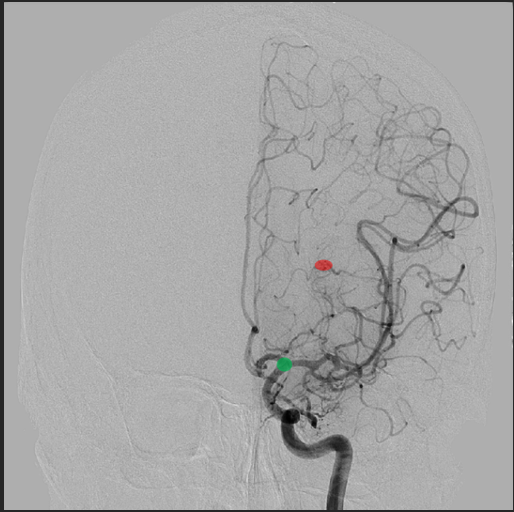
Op de sagittale en coronale DSA werd door een ervaren radioloog en neurochirurg de locatie van de AVM bepaald. Ook werd de bifurcatie, waar de a. carotis interna splitste in de a. cerebri media en a. cerebri anterior gelokaliseerd (Figuur 4). Deze werden later gebruikt voor de validatie.
Registratie
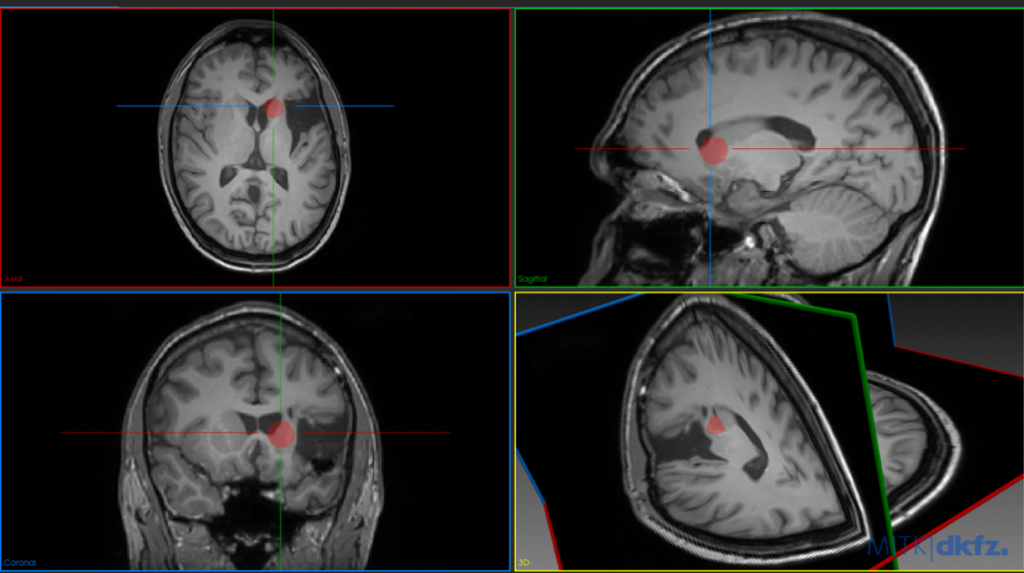
Met behulp van de Elastix-software werd een transformatie toegepast op de DSA-segmentaties, zodat deze zo goed mogelijk gematcht werd met de corresponderende MRI-segmentaties. Hiervoor werd een similarity-transformatie gebruikt, die de DSA kan roteren, transleren en vergroten of verkleinen, maar waarbij geen non-rigide vervorming van het beeld mogelijk is (Figuur 5). De meest optimale transformatie werd ook toegepast op een leeg beeld dat alleen de locaties van de AVM en de bifurcatie bevatte, zodat ook de nieuwe locaties van deze punten over de MRI werden geprojecteerd.

Validatie
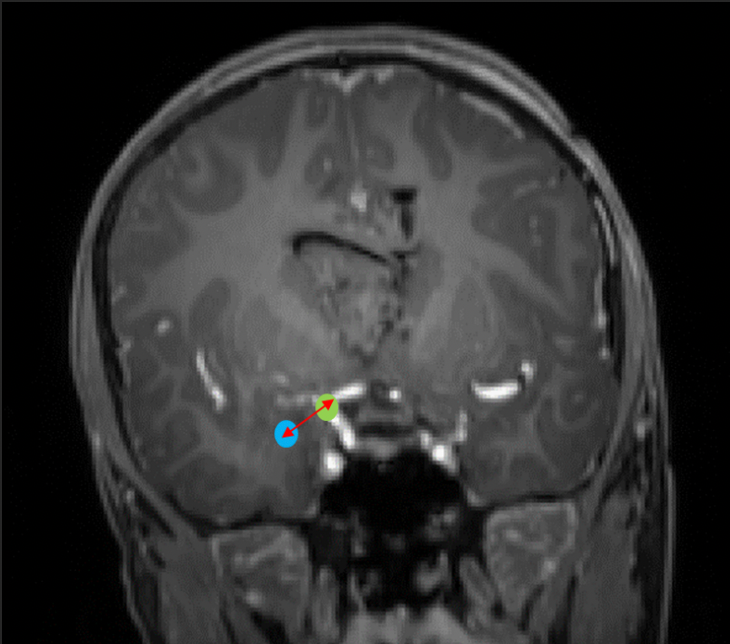
De afstand tussen de nieuwe locatie van de bifurcatie van de a. carotis interna werd vergeleken met de daadwerkelijke locatie, die zichtbaar was op de MRI. De afstand tussen deze punten geeft een goed beeld van de kwaliteit van de registratie en is een representatieve maat voor de nauwkeurigheid van de geschatte locatie van de AVM (Figuur 6).
Resultaten
De data van zeven patiënten (Leids Universitair Medisch Centrum), die bekend waren met een mAVM werd met toestemming van de wetenschapscommissie radiologie geanonimiseerd verkregen uit het elektronisch patiëntendossier. Deze data bestond uit de meest recente DSA waarop de mAVM zichtbaar was volgens het verslag van de radioloog. Ook werd de laatste T1-gewogen MRI met gadolinium-contrast die voor deze DSA verkregen was geanalyseerd.
Na de registratie van de DSA-segmentatie op de MRI-segmentatie werd de nieuwe geschatte locatie van de bifurcatie van de a. carotis interna vergeleken met de geobserveerde werkelijke locatie van dit anatomische punt op de MRI. In de tabel is de afstand tussen deze punten (in mm) weergegeven. Deze fout in de schatting van de locatie varieerde van 6 tot 24 mm met een gemiddegemiddelde van 13,43 mm en een standaarddeviatie van 6,00 mm.

Discussie
Voor deze studie is een software-tool ontwikkeld die de 3D locatie op een MRI van een AVM kan berekenen op basis van de coördinaten van de AVM op een coronale en sagittale DSA. Bij validatie bleek de tool een redelijke nauwkeurigheid te geven, met een gemiddelde fout van 13,43 mm en een standaarddeviatie van 6,00 mm. Hoewel deze waardes te hoog zijn om echt bij te dragen aan nauwkeurigere en efficiëntere preoperatieve planning, zijn de resultaten interessant genoeg om dit onderzoek te vervolgen.
Limitaties
De ligging van het hoofd tijdens de opname van de DSA is niet exact bepaald. Hoewel er gecorrigeerd wordt voor de hoek van de C-boog tijdens de acquisitie van de DSA, wordt er geen rekening gehouden met de hoek van het hoofd tijdens deze opname. Omdat er alleen gekeken wordt naar de hoek van de C-boog ten opzichte van de tafel is het mogelijk dat de MRI ook na de rotatie nog niet in hetzelfde assenstelsel gedraaid is als de DSA. Dit kan leiden tot fouten in de projectie van de AVM. Dit probleem zou opgelost kunnen worden door ook de hoek van de patiënt met de tafel te meten.
Een andere limitatie is de mismatch in het field of view van de DSA in vergelijking met de MRI. Op de sagittale DSA wordt alleen het achterste deel van het hoofd gevisualiseerd, en loopt de opname inferior bijna tot de nek. Op de MRI daarentegen zijn alleen de gehele hersenen gevisualiseerd. Dit verschil in field of view leidt tot fouten in de registratie en daardoor een minder nauwkeurige projectie van de AVM.
Als laatste wordt bij de segmentatie van het hoofd op de DSA gebruikt gemaakt van een smoothing-algoritme. Deze operatie voorkomt ruis in de segmentatie, maar heeft ook het verlies van details tot gevolg. Deze verminderde detaillering kan leiden tot een minder accurate registratie.
Toekomstig onderzoek
Een interessant vooruitzicht is het projecteren van de gehele vasculatuur van de hersenen op de MRI. Hiervoor moet eerst een 3D reconstructie worden gemaakt van de vaten aan de hand van de sagittale en coronale DSA. Vervolgens moet deze reconstructie geregistreerd worden op de MRI. Hiervoor is echter wel betere segmentatie nodig van de vaten op de MRI, en zou bijvoorbeeld MRA gebruikt moeten worden.
Ook is een belangrijke volgende stap het uitwerken van de software tot een gebruiksvriendelijke app, die in de kliniek gebruikt kan worden. Hiervoor moet de software geïntegreerd worden in een graphical user interface (GUI) met minimale gebruikersinteractie. Een andere optie is om een koppeling te maken met het Picture Archiving and Communication System (PACS), zodat de fusie tussen MRI en DSA direct in deze interface kan plaatsvinden.
Referenties
[1] Bokhari MR, Bokhari SRA. Arteriovenous Malformation Of The Brain. [Updated 2021 Dec 12]. In: StatPearls [Internet]. Treasure Island (FL): StatPearls Publishing; 2022 Jan-. Available from: https://www.ncbi.nlm.nih.gov/books/NBK430744/
[2] Cellerini M, Mangiafico S, Villa G, Nistri M, Pandolfo C, Noubari H, et al. Cerebral microarteriovenous malformations: Diagnostic and therapeutic features in a series of patients. Am J Neuroradiol. 2002;23(6):945–52.
[3] Alen JF, Lagares A, Paredes I, Campollo J, Navia P, Ramos A, et al. Cerebral microarteriovenous malformations: A series of 28 cases. J Neurosurg. 2013;119(3):594–602.
[4] Gross BA, Frerichs KU, Du R. Sensitivity of CT angiography, T2-weighted MRI, and magnetic resonance angiography in detecting cerebral arteriovenous malformations and associated aneurysms. J Clin Neurosci [Internet]. 2012;19(8):1093–5. Available from: http://dx.doi.org/10.1016/j.jocn.2011.11.021
[5] Ide S, Hirai T, Morioka M, Kai Y, Yano S, Kawano T, et al. Usefulness of 3D DSA-MR Fusion Imaging in the Pretreatment Evaluation of Brain Arteriovenous Malformations. Acad Radiol [Internet]. 2012;19(11):1345–52. Available from: http://dx.doi.org/10.1016/j.acra.2012.07.001
[6] Tranvinh E, Heit J, Hacein-Bey L, Provenzale J, Wintermark M. Contemporary Imaging of Cerebral Arteriovenous Malformations. American Journal of Roentgenology. 2017;208(6):1320-1330.
[7] Cenzato M, Dones F, Boeris D, Marcati E, Fratianni A, Crisa F et al. Contemporary tools in arteriovenous malformations surgery. Journal of Neurosurgical Sciences. 2018;62(4).
[8] Lemieux L, Jagoe R, Fish D, Kitchen N, Thomas D. A patient-to-computed-tomography image registration method based on digitally reconstructed radiographs. Medical Physics. 1994;21(11):1749-1760.
[9] Gottesfeld Brown LM, Boult TE. Registration of planar film radiographs with computed tomography. Proceedings of the Workshop on Mathematical Methods in Biomedical Image Analysis. 1996;
[10] Klein S, Staring M, Murphy K, Viergever M a., Pluim J. Elastix: A Toolbox for Intensity-Based Medical Image Registration. IEEE Trans Med Imaging. 2010;29(1):196–205.
[11] Patiëntfolder ‘Neurointerventie AVM of Tumor’, Afdeling Radiologie Erasmus MC